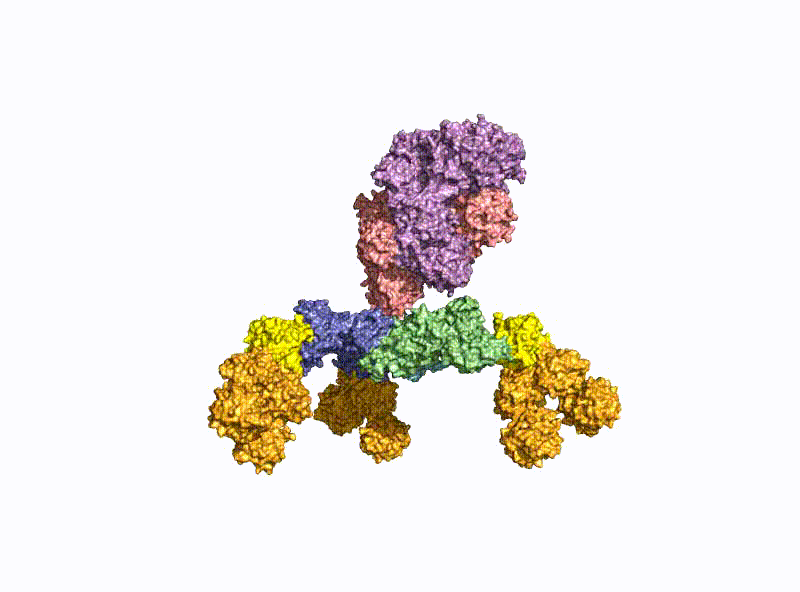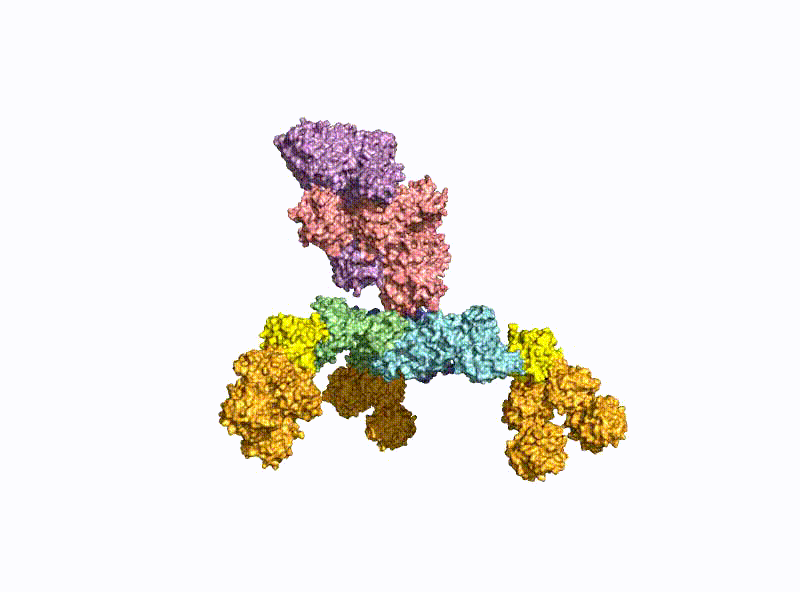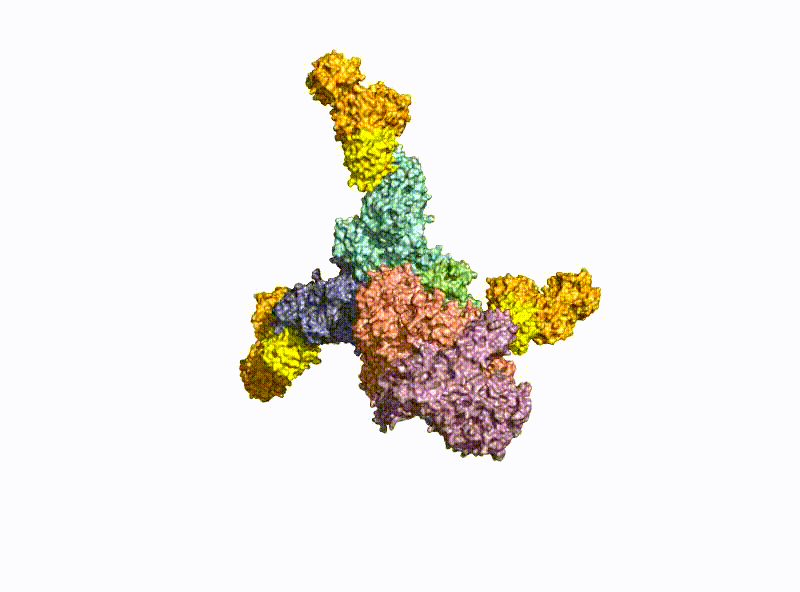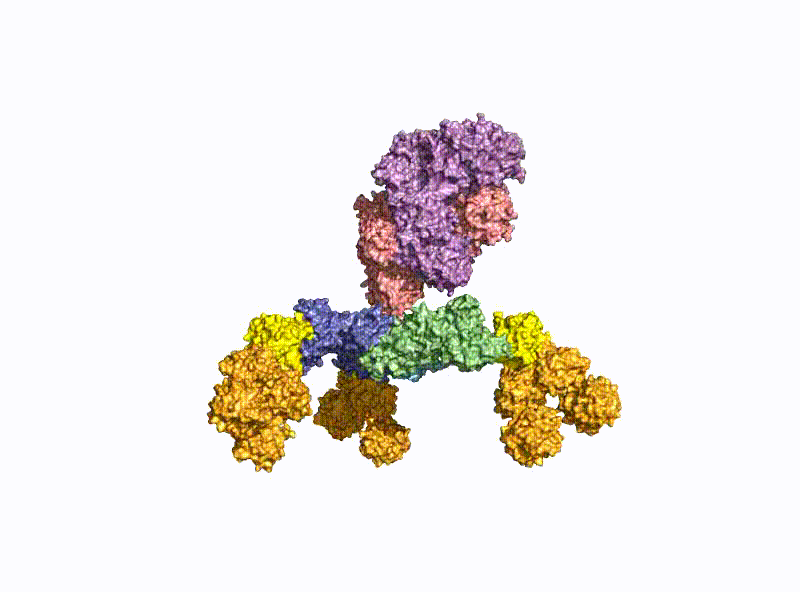Mitglieder der Arbeitsgruppe Rummel

von links nach rechts:
Dr. rer. nat. Jasmin Weisemann E-Mail
Melvin Bönninger (M.Sc.Biomedizin) E-Mail
Dr. rer. nat. Andreas Rummel E-Mail
Urs von der Kammer (M.Sc.Biochemie) E-Mail
ohne Foto:
Nadja Krez E-Mail
Ehemalige Mitglieder der Arbeitsgruppe ohne Foto::
Dr. rer. nat. Jana Marielle Modenbach
Dr. Adina Przykopanski
Denise Bellmann (M.Sc.Applied Biology)
Ariane Piwek (M.Sc.Biomedizin)
Forschung

Der Arbeitsgruppenleiter Dr. Andreas Rummel erforscht seit 1998 kontinuierlich grundlegende Wirkmechanismen von clostridiellen Neurotoxinen an der Medizinischen Hochschule Hannover (MHH) und hat so dazu beigetragen, das Wissen über die molekularen Wirkmechanismen von Botulinum Neurotoxinen (BoNTs) sowie deren Detektion zu vergrößern [1, 2]. Hierdurch hat er derzeit 60 peer-reviewed Originalveröffentlichungen und 13 Übersichtsartikel / Buchkapitel in z. T. höchstrangigen Fachzeitschriften publiziert, welche insgesamt über 6000-mal zitiert wurden (Google Scholar Stand 2026). Am Institut für Toxikologie der MHH hat er mit seiner Arbeitsgruppe eine hohe Expertise auf dem Gebiet der rekombinanten Expression von BoNTs in E. coli aufgebaut. Dr. Rummel war bzw. ist u.a. Antragssteller und Projektleiter in den vom BMBF/BMFTR geförderten Projekten FuMiBoNT(Fk 031A212A; Abschlussbericht), SensTox (Fk 13N13794; Abschlussbericht), TiViBoNT (Fk 031L0111B/161L0111B; Abschlussbericht) und ImpliBONT (Fk 16LW0295/03LW0295). In diesen wurden erfolgreich Methoden zur Herstellung von bakteriellen Proteintoxinen und deren Proteinrezeptoren erforscht, welche als Reagenzien zum Nachweis u.a. von Botulinum Neurotoxin in einer Tierversuchsersatzmethode zur Botulismus-Diagnostik eingesetzt werden. Des Weiteren war Dr. Rummel Sprecher des deutsch-franzöischen X-BAT Konsortiums, welches von ANR und BMFTR (Fk 13N15512) von 2021-2025 gefördert wurde und die Entwicklung eines decavalenten Botulism Antitoxins zum Ziel hat. Außerdem betreibt die AG Rummel erfolgreich eine Tierversuchsersatzmethode zum Nachweis von BoNT, den Nervus phrenicus Hemidiaphragma Assay [3], welcher u.a. auch in der Lehre zur Untersuchung der Wirkung von Muskelrelaxantien eingesetzt wird (Abb. 1).
BoNT
Botulinum Neurotoxine
Unter der Bezeichnung Botulinum Neurotoxin (BoNT) verbirgt sich eine Familie von über 50 bakteriellen Proteintoxinen, welche die Acetylcholinfreisetzung in der neuromuskulären Endplatte blockieren und so im Falle einer oralen Intoxikation die tödlich verlaufende Krankheit Botulismus hervorrufen. Von 1897 bis 1970 wurden sieben Serotypen BoNT/A-G identifiziert, welche Unterschiede in der Aminosäuresequenz von 37.2%–69.6% aufweisen [4]. Von diesen wurden bisher BoNT/A und B als Muskelrelaxantien zugelassen, obwohl auch BoNT/C, D, E und F im Menschen pharmakologisch wirksam sind [5, 6]. Im Oktober 2013 wurde ein neuer BoNT Serotyp HA, welcher von einem bivalenten C. botulinum-Stamm, isoliert aus dem Stuhl eines Patienten mit Säuglingsbotulismus, produziert wird, identifiziert [7, 8, 9]. Mittlerweile wurden weitere Toxingene kodierend neue Serotypen, provisorisch BoNT/X, eBoNT/J aka BoNT/En genannt, identifiziert und mittels nichttoxischer Fragmente zum Teil molekularbiologisch und strukturell charakterisiert wurde [10, 11, 12, 13, 14].
Kürzlich wurde das PMP1, ein BoNT-ähnliches Neurotoxin, das selektiv auf Anopheles-Mücken abzielt, aus Paraclostridium bifermentans-Stämmen, die in endemischen Anophelesgebieten auf zwei Kontinenten gesammelt wurden, identifiziert [15].
Mit einem Molekulargewicht von 150 kDa sind die BoNTs klassische AB-Toxine (Abb. 2) und bestehen aus einer leichten Kette (LC) mit 50 kDa und einer schweren Kette (HC) mit 100 kDa, welche über eine Disulfidbrücke kovalent miteinander verknüpft sind. Die HC unterteilt sich in eine Translokationsdomäne HN und eine Rezeptorbindungsdomäne HC mit jeweils 50 kDa.
Wissenschaftliche Schwerpunkte
Ein Schwerpunkt unserer AG war und ist die Aufklärung der Rezeptorstrukturen, ihrer Bindungstaschen in HC sowie die Nutzbarmachung dieses Wissens zur Entwicklung von pharmazeutisch verbesserten Wirkstoffen, Fängerstrukturen zur Diagnostik von BoNT und Inhibitoren zur Behandlung bzw. Verhinderung eines Botulismus.
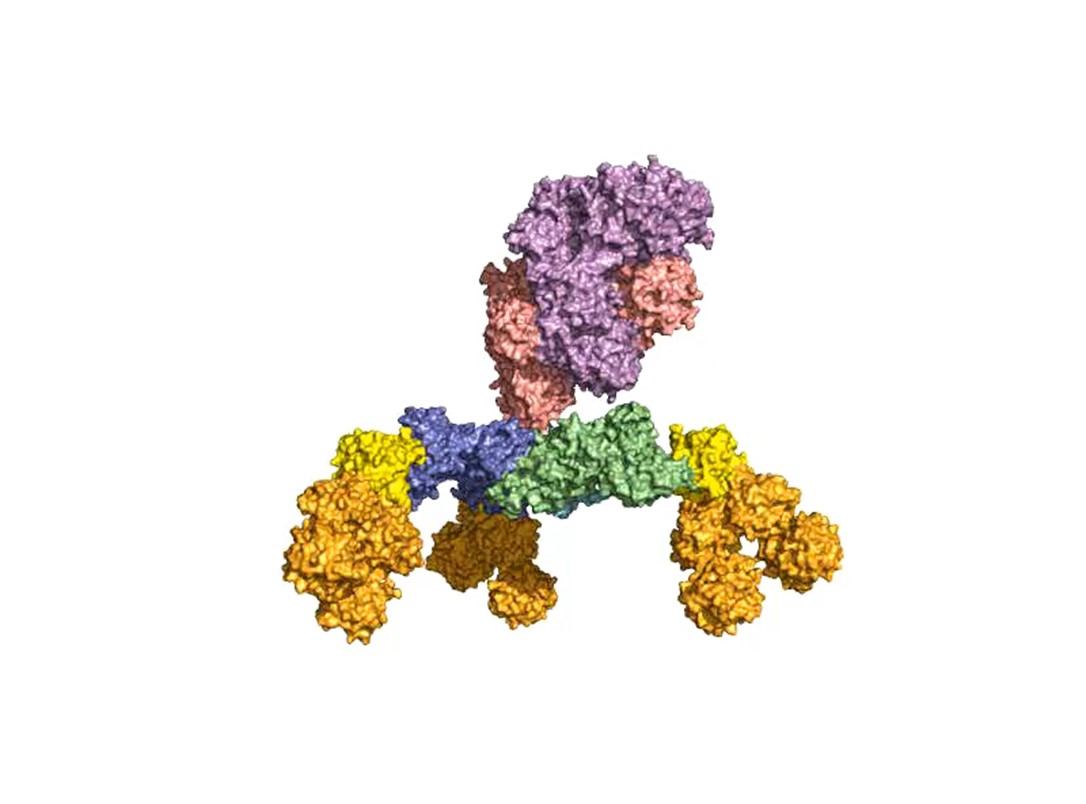
Ein zweiter Schwerpunkt ist die Erforschung der oralen Aufnahme von BoNTs, welche als einige der wenigen Proteine die harschen Bedingungen im Magen-Darm-Trakt unbeschadet überstehen. Hierzu bilden sie mit dem nahezu gleich großen Nichttoxischen Nichthämagglutinin (NTNHA) pH-abhängig einen säure- und proteasestabilen mittelgroßen (M-) Komplex (Abb. 3), welchen die AG Rummel 2012 gemeinsam mit der AG R. Jin (UCI, CA) strukturell aufklären und in Science publizieren konnte [18]. Drei weitere Hämagglutinine (HA) unterschiedlicher Größe bilden den sogenannten HA-Komplex von 510 kDa, welcher sich mit dem M-Komplex zu dem 760 kDa großen BoNT (L-) Komplexe verbindet, der damit ein hochpotentes Nahrungsmittelgift darstellt. Unsere AG erforschte im Detail den Aufbau des L-Komplexes und die Interaktion des HA-Komplexes mit Zuckerstrukturen im Dünndarm [17] sowie dessen intestinalen Aufnahmemechanismus durch Aufbruch von Zell-Zell-Kontakten [19].
Zukünftige Forschungsfelder liegen im Bereich der molekularen Charakterisierung von neuen Mitglieder der BoNT-Familie in Bezug auf orale Aufnahme und intestinale Resorption, neuronale Rezeptorinteraktion und enzymatische Wirkung in der Nervenzelle.
Hierzu sind wir stets auf der Suche nach interessierten Bachelor- und Master-Studenten aus naturwissenschaftlichen Studiengängen sowie Doktoranden im StrucMed-Programm.
Kontakt:
Dr. Andreas Rummel
E-Mail
Literatur
- [1] Rummel, A., The long journey of botulinum neurotoxins into the synapse. Toxicon, 2015. 107(Pt A): p. 9-24.
- [2] Rummel, A., Two Feet on the Membrane: Uptake of Clostridial Neurotoxins. Curr Top Microbiol Immunol, 2016.
- [3] Bigalke, H. and A. Rummel, Botulinum Neurotoxins: Qualitative and Quantitative Analysis Using the Mouse Phrenic Nerve Hemidiaphragm Assay (MPN). Toxins (Basel), 2015. 7(12): p. 4895-905.
- [4] Hill, K.K. and T.J. Smith, Genetic diversity within Clostridium botulinum serotypes, botulinum neurotoxin gene clusters and toxin subtypes. Curr Top Microbiol Immunol, 2013. 364: p. 1-20.
- [5] Montecucco, C. and J. Molgo, Botulinal neurotoxins: revival of an old killer. Curr Opin Pharmacol, 2005. 5(3): p. 274-9.
- [6] Kutschenko, A., et al., Botulinum neurotoxin serotype D - A potential treatment alternative for BoNT/A and B non-responding patients. Clin Neurophysiol, 2019. 130(6): p. 1066-1073.
- [7] Barash, J.R. and S.S. Arnon, A novel strain of Clostridium botulinum that produces type B and type H botulinum toxins. J Infect Dis, 2014. 209(2): p. 183-91.
- [8] Dover, N., et al., Molecular characterization of a novel botulinum neurotoxin type H gene. J Infect Dis, 2014. 209(2): p. 192-202.
- [9] Gonzalez-Escalona, N., et al., Draft Genome Sequence of Bivalent Clostridium botulinum Strain IBCA10-7060, Encoding Botulinum Neurotoxin B and a New FA Mosaic Type. Genome Announc, 2014. 2(6).
- [10] Zhang, S., et al., Identification and characterization of a novel botulinum neurotoxin. Nat Commun, 2017. 8: p. 14130.
- [11] Masuyer, G., et al., Structural characterisation of the catalytic domain of botulinum neurotoxin X - high activity and unique substrate specificity. Sci Rep, 2018. 8(1): p. 4518.
- [12] Williamson, C.H.D., et al., Botulinum-neurotoxin-like sequences identified from an Enterococcus sp. genome assembly. bioRxiv, 2017: p. 228098.
- [13] Brunt, J., et al., Identification of a novel botulinum neurotoxin gene cluster in Enterococcus. FEBS Lett, 2018.
- [14] Zhang, S., et al., Identification of a Botulinum Neurotoxin-like Toxin in a Commensal Strain of Enterococcus faecium. Cell Host Microbe, 2018. 23(2): p. 169-176 e6.
- [15] Contreras, E., et al., A neurotoxin that specifically targets Anopheles mosquitoes. Nat Commun, 2019. 10(1): p. 2869.
- [16] Brunger, A.T. and A. Rummel, Receptor and substrate interactions of clostridial neurotoxins. Toxicon, 2009. 54(5): p. 550-60.
- [17] Lee, K., et al., Structure of a bimodular botulinum neurotoxin complex provides insights into its oral toxicity. PLoS Pathog, 2013. 9(10): p. e1003690.
- [18] Gu, S., et al., Botulinum neurotoxin is shielded by NTNHA in an interlocked complex. Science, 2012. 335(6071): p. 977-81.
- [19] Lee, K., et al., Molecular basis for disruption of E-cadherin adhesion by botulinum neurotoxin A complex. Science, 2014. 344(6190): p. 1405-10.