Krankheitsbezug der Forschung
Die übergeordnete Forschungsrichtung des Instituts für Pharmakologie ist die molekulare und pathophysiologische Analyse von Signaltransduktionsvorgängen.
- Immunologische Erkrankungen:
Colitis ulcerosa, Morbus Crohn - Bakterielle Infektionen:
Pseudomonas aeruginosa-Infektionen - Tumorerkrankungen:
Mammakarzinom, Colonkarzinom, Kopf-/Hals-Karzinome
Aktuelle Forschungsprojekte
Was wir wissen:
Identifizierung von Targetmolekülen und erste Ergebnisse der Funktion
Neben den kanonischen zyklischen Moleküle, den bekannten Second Messenger cAMP und cGMP, gibt es auch die nicht-kanonischen zyklischen Moleküle cCMP und cUMP. cAMP und cGMP haben vielfältige Wirkungen auf unterschiedliche Funktionen von Zellen, wie Proliferation, Genexpressionsveränderung oder auch Induktion von Apoptose. Viele dieser Mechanismen werden über die Aktivierung von PKA durch cAMP und von PKG durch cGMP vermittelt.
Im Institut für Pharmakologie werden seit einigen Jahren verschiedene Untersuchungen zu cCMP und cUMP durchgeführt. Es gelang der erfolgreiche Nachweis sowohl von cCMP als auch cUMP in verschiedenen Zelllinien, Bakterien und Organen unterschiedlichster Spezies durch die im Institut etablierte MS-Analytik der Research Core Unit Metabolomics. In mehreren Publikationen konnte gezeigt werden, dass cCMP und cUMP an PKA und PKG binden und diese Kinasen auch aktivieren können. Von anderen Arbeitsgruppen wurden weitere cCMP-Targets wie EPAC (exchange factor directly activated by cAMP) und die Ionenkanäle HCN 2 und HCN 4 gefunden. Auch wir haben bereits weitere cCMP- und cUMP-bindende Proteine nachweisen können. Außerdem konnten cCMP- und cUMP-generierende als auch -abbauende Proteine (Phosphodiesterasen, PDEs) identifiziert werden.
In verschiedenen Zelllinien konnte gezeigt werden, dass cCMP und cUMP Apoptose induzieren können. In speziellen Zellen (S49 kin-) erfolgte der Nachweis, dass dazu alle bisher bekannten Bindungspartner nicht nötig sind, was bedeutet, dass es weitere Targetmoleküle geben muss (s. Abb.)

In anderen Zelllinien (z. B. K562), die resistent gegen cCMP- und cUMP-induzierter Apoptose waren, konnte diese durch Inhibierung spezieller Transporter ebenfalls ausgelöst werden.
In aktuellen Untersuchungen werden die Effekte von cCMP und cUMP in Mammakarzinom-Zelllinien analysiert. Beim Mammakarzinom findet man unterschiedliche Formen, die sich hauptsächlich durch die Expression der Rezeptoren für Östrogene und Progesteron unterscheiden. Werden diese Rezeptoren kaum oder gar nicht exprimiert, gilt diese Form der Erkrankung als schwer therapierbar. Eine Hemmung der Proliferation konnte in einer Zelllinie, die als Modell für diese schwer zu therapierende Form verwendet wird, durch Inkubation mit cCMP und auch cUMP erreicht werden. Durch Kombination von Efflux-Inhibitoren und speziellen PDE-Inhibitoren konnte dieser Effekt weiter verstärkt werden.
Aktuelle Forschungsprojekte:
Möglichkeiten von Laborpraktika oder Bachelor- bzw. Masterarbeiten
- Analyse der Proliferationshemmung in den Mammakarzinom-Zelllinien, weiterer Zelllinien oder primärer Zellen, Untersuchungen zum Wirkmechanismus
- Identifizierung weiterer cCMP- und cUMP-bindender Proteine mittels cNMP-Agarosen oder Biotin-cNMPs
- Untersuchungen zu den neu identifizierten cCMP- und cUMP-bindenen Proteinen, z.B. die Charakterisierung der Bindung von cCMP und cUMP an AKAPs
Nähere Informationen:
Dr. Sabine Wolter, E-Mail
Ausgewählte Publikationen zu den Forschungsprojekten:
Seifert R, Bugert JJ. cCMP and cUMP phosphodiesterases in viral infections. Trends Biochem Sci. 2023; 48: 835-838.
Seifert R, Schirmer B. cCMP and cUMP come into the spotlight, finally. Trends Biochem Sci. 2022; 47: 461-463.
Wolter S, Dittmar F, Seifert R. cCMP and cUMP in Apoptosis: Concepts and Methods. Handb Exp Pharmacol 2017; 238: 25-47
Wolter S, Kloth C, Golombek M, Dittmar F, Försterling L, Seifert R. cCMP causes caspase-dependent apoptosis in mouse lymphoma cell lines. Biochem Pharmacol 2015;98: 119-131
Hammerschmidt A, Chatterji B, Zeiser J, Schröder A, Genieser HG, Pich A, Kaever V, Schwede F, Wolter S, Seifert R. Binding of Regulatory Subunits of Cyclic AMP-Dependent Protein Kinase to Cyclic CMP Agarose. PLoS One. 2012; 7: e39848
Wolter S, Golombek M, Seifert R. Differential activation of cAMP- and cGMP-dependent protein kinases by cyclic purine and pyrimidine nucleotides. Biochem Biophys Res Commun 2011; 415: 563-566
Signaling via cCMP and cUMP - a short overview and appetizer for the research community, particularly prospective students,
eine Zusammenfassung als pdf zum Download von Prof. Seifert
Viele bakterielle Toxine weisen eine Nukleotidylzyklaseaktivität auf und greifen so in die Signaltransduktion infizierter Wirtszellen ein. So wird z.B. das Effektorprotein ExoY durch das nadelartige Typ-3-Sekretionssystem (T3SS) von Pseudomonas (P.) aeruginosa in Wirtszellen eingeschleust und synthetisiert dort neben cAMP und cGMP auch die nicht-kanonischen zyklischen Pyrimidinnukleotide cUMP und cCMP - in der Tat sind sogar je nach Infektionsmodell cUMP bzw. cGMP die mengenmäßig meistproduzierten zyklischen Nukleotide.
ExoY benötigt zur Aktivierung der Zyklaseaktivität F-Aktin als zytosolischen Kofaktor. Es ist bisher aber nicht bekannt, ob Aktin der einzige Kofaktor für ExoY ist, da bspw. die cCMP- und cUMP-Produktion von in vitro mit Aktin inkubiertem ExoY nicht untersucht worden ist.
Wir führen eine vollständige enzymatische Charakterisierung des Enzyms in Bezug auf die verschiedenen Substrate durch, um die Determinanten der cUMP- bzw. cCMP-Produktion zu ermitteln. Dies ist umso wichtiger, da relativ neue Daten belegen, dass katalytisch aktives ExoY in der Lage ist, die angeborene Immunantwort zu hemmen, aber völlig unklar ist, welches der zyklischen Nukleotide dafür verantwortlich ist. Es fehlt hier bisher die Möglichkeit, dies durch Anwendung anderer natürlicher, bakterieller Toxine zu untersuchen, da es schlicht keine bekannten Toxine gibt, die präferentiell cUMP oder cCMP produzieren. Die Struktur von ExoY wurde erst vor kurzem zum Großteil aufgeklärt. Analysen gezielter ExoY-Mutationen fehlen aber noch völlig, daher generieren wir durch gerichtete Evolution (site-directed / random mutagenesis) zur Zeit Effektorprotein-Mutanten, die zukünftig zur Erforschung von cUMP- und cCMP-Funktionen in Säugerzellen eingesetzt werden können. Das Screening bereits zugelassener Arzneimittel hinsichtlich ihrer Wirkung auf die Nukleotidylzyklaseaktivität von ExoY komplementiert diese Vorgehensweise.
Wir wollen auch die molekularen Targets und die Funktion der zyklischen Pyrimidinnukleotide in Zellen des angeborenen und adaptiven Immunsystems identifizieren und so die Signaltransduktion der nicht-kanonischen Nukleotide und deren funktionelle Rolle bei der antiinfektiven Immunantwort charakterisieren.
Mögliche Themen für experimentelle Arbeiten
(Bachelor- und Masterarbeiten, Laborpraktika):
- Charakterisierung der Signaltransduktion identifizierter 3‘,5‘-cUMP targets
- Metabolomisches Screening nach cNMP Effektor-Signalwegen in Lymphozyten
- Screening nach Wirkstoffen, die die Nukleotidylzyklaseaktivität von ExoY modulieren
- Analyse der T-Zell-Toxizität des Effektorproteins ExoY aus P. aeruginosa
Methodenspektrum
Zusätzlich zu den grundlegenden molekularbiologischen, immunologischen, (protein)biochemischen und zellbiologischen Labortechniken:
- Mikroskopie (Epifluoreszenz / konventionelle Lichtmikroskopie)
- Durchflusszytometrie
- Multiplex bead-based ELISA
- Zellseparation mittels magnetgebundener Antikörper
- High-throughput Spektralphotometrie/-fluorimetrie/-luminometrie
- ELISA, FRET, BRET
- Küvetten-Spektralfluorimetrie
- Quantitative Real-time PCR
- Western-Blot (ECL)
- Chromatographische Präparation und Analyse von Proteinen (ÄKTA)
- Real-time Bioimpedanzmessung
- LC-IMS/QTOF, LC-MS/MS, GC-MS/MS (Kooperation mit RCU Metabolomics)
Ausgewählte Pubklikationen:
Bähre H, Hartwig C, Munder A, Wolter S, Stelzer T, Schirmer B, Beckert U, Frank DW, Tümmler B, Kaever V, Seifert R. cCMP and cUMP occur in vivo. Biochem Biophys Res Commun 2015; 460: 909-914
https://doi.org/10.1016/j.bbrc.2015.03.115
Seifert, R, Schneider, EH, Bähre H. (2015). From canonical to non-canonical cyclic nucleotides as second messengers: pharmacological implications. Pharmacology & therapeutics, 148, 154–184. https://doi.org/10.1016/j.pharmthera.2014.12.002
Seifert R. (2015). cCMP and cUMP: emerging second messengers. Trends in biochemical sciences, 40(1), 8–15. https://doi.org/10.1016/j.tibs.2014.10.008
Seifert R. (2017). cCMP and cUMP Across the Tree of Life: From cCMP and cUMP Generators to cCMP- and cUMP-Regulated Cell Functions. Handbook of experimental pharmacology, 238, 3–23. https://doi.org/10.1007/164_2016_5005
Munder A, Rothschuh J, Schirmer B, Klockgether J, Kaever V, Tümmler B, Seifert R, Kloth C. The Pseudomonas aeruginosa ExoY phenotype of high-copy-number recombinants is not detectable in natural isolates. Open Biol 2018; 8: pii: 170250.
https://doi.org/10.1098/rsob.170250
Kloth, C., Schirmer, B., Munder, A., Stelzer, T., Rothschuh, J., & Seifert, R. (2018). The role of Pseudomonas aeruginosa ExoY in an acute mouse lung infection model. Toxins 10(5), 185
https://doi.org/10.3390/toxins10050185
Nähere Informationen
Dr. Bastian Schirmer, E-Mail
Histamin [2-(4-Imidazolyl)-ethylamin; β-Imidazolylethylamin] ist ein endogenes biogenes Amin, das in allen Säugetiergeweben vorkommen kann. Es wird durch Decarboxylierung der Aminosäure L-Histidin mittels der Histidin-Decarboxylase (HDC) synthetisiert und durch Methylierung über die Histamin-N-Methyltransferase zu N-Methylhistamin oder oxidative Desaminierung über die Diaminoxidase zu Imidazolessigsäure abgebaut.
Als schon Anfang des 20. Jahrhunderts charakterisierter Botenstoff, der bei inflammatorischen Prozessen eine Rolle spielt, ist Histamin einer der am besten untersuchten Mediatoren in der Biomedizin. Histamin hat pleiotrope Wirkungen im gesunden und kranken Organismus, indem es vier Subtypen von GPCRs der Klasse A aktiviert, die chronologisch in der Reihenfolge ihrer Entdeckung als Histamin H1-Rezeptor (H1R), H2R, H3R und H4R benannt wurden. Die Histamin-Rezeptorsubtypen weisen unterschiedliche Affinität für Histamin auf, weshalb neben der Zelltyp-spezifischen Expression der Rezeptorsubtypen lokale Histaminkonzentrationen wichtige Determinanten der biologischen Reaktion sind.
Das Hauptinteresse am Institut für Pharmakologie gilt dem H4R, der inflammatorische und immunologische Prozesse beeinflusst. Mithilfe von genetisch veränderten Mäusen (H4R-KO) und selektiven pharmakologischen Werkzeugen (Agonisten und Antagonisten) untersuchen wir z.Zt. die Funktion des H4R in vivo und in vitro in Modellen der entzündlichen Darmerkrankung.
Eine experimentelle akute Colitis in Mäusen kann durch genetische oder pharmakologische Blockade der H4R Funktion abgemildert werden. Daher gehen wir davon aus, dass der H4R bei der Pathogenese der Colitis eine Rolle spielt. Da wir bisher diese aber nicht ausreichend verstehen, stellt ihre Entschlüsselung aktuell den Fokus unserer Arbeit dar.
Die Funktion des H4R in der Colitis untersuchen wir auf zellulärer Ebene. Unser Hauptaugenmerk gilt dabei den Epithelzellen, die den H4R funktionell exprimieren. Wir untersuchen die Beeinflussung der epithelialen Barriere durch Histamin, was zur Ausprägung der Colitis beitragen kann. Eine der wesentlichen Komplikation der Colitis ist die Entwicklung des Coloncarcinoms. Auch in seiner Pathogenese spielt Histamin und der H4R eine Rolle, die wir aufklären wollen.

Aktuelle Projekte, die Möglichkeiten für Praktika und Bachelor-/Masterarbeiten bieten:
- Herstellung von permanenten Epithelzellkulturen zur Untersuchung der H4R-Funktion.
- MS-basierte Analyse der der Änderung des Metaboloms durch die Stimulation mit Histamin
- MS-basierte Messung der Histamin- bzw. Histaminmetabolitenkonzentration: Vergleich von Proben aus gesunden und erkrankten Modellen
- Analyse der Funktion des H4R auf an der Colitis bzw. dem Coloncarcinom beteiligten Zellen des Immunsystems
- Entwicklung von 3-dimensionalen in vitro Modellen zu funktionellen Untersuchung der Colitis und des Coloncarcinoms
- Analyse möglicher Histamin Rezeptor-Komplexe: homo/hetero Di-/Polymere
- Analyse einer Interaktion von Histamin Rezeptoren mit GIRK1/2
Ausgewählte Publikationen
C. Hartwig, A. Munder, S. Glage, D.Wedekind, H. Schenk, R. Seifert, and D. Neumann
The histamine H4-receptor (H4R) regulates eosinophilic inflammation in ovalbumin-induced experimental allergic asthma in mice. Eur. J. Immunol., 45:1129-1140 (2015).
B. Schirmer, T. Rezniczek, R. Seifert, and D. Neumann. Proinflammatory role of the histamine H4-receptor in dextrane sodium sulphate-induced acute colitis. Biochem. Pharmacol., 98:102-109 (2015).
S. Beermann, G. Bernhardt, A. Buschauer, R. Seifert, and D. Neumann
Histamine H1- and H4-receptor signaling cooperatively regulate MAPK activation
Biochem. Pharmacol., 98:432-439 (2015).
E.J. Wunschel, B. Schirmer, R. Seifert, and D. Neumann
Lack of histamine H4-receptor expression aggravates TNBS-induced colitis in mice
Frontiers Pharmacol. 8:642 (2017).
B. Schirmer, L. Bringmann, R. Seifert, and D. Neumann
In vivo evidence for partial activation of eosinophils via the histamine H4-receptor: adoptive transfer experiments using eosinophils from H4R-/- and H4R+/+ mice
Frontiers Immunol. 9:2119 (2018).
B. Schirmer, L. Lindemann, K.S. Bittkau, R. Isaev, D. Bösche, M. Juchem, R. Seifert, and D. Neumann
Mouse colonic epithelial cells functionally express the histamine H4 receptor
J. Pharmacol. Exp. Ther., 373:167-174 (2020).
B. Schirmer, T. Rother, I. Büsch, A. Bleich, C. Werlein, D. Jonigk, R. Seifert, and D. Neumann
Genetic deficiency of the histamine H4 receptor reduces experimental colorectal carcinoma in mice
Cancers, 12:912 (2020).
Schirmer B, Neumann D. The Function of the Histamine H4 Receptor in Inflammatory and Inflammation-Associated Diseases of the Gut. Int J Mol Sci 2021; 22: 6116. doi: 10.3390/ijms22116116.
Schrammel JC, König M, Frommer M, Andersen KS, Kirsten M, Seifert R, Neumann D, Schirmer B. Histamine H1- and H4-receptor expression in human colon-derived cell lines. Naunyn Schmiedebergs Arch Pharmacol 2023; 396:3683-3693. doi: 10.1007/s00210-023-02565-8.
Nähere Informationen:
Prof. Dr. Detlef Neumann, E-Mail
Viren und ihre Wirte befinden sich in einem steten evolutionären Wettrennen. In Wirtsorgansimen wie Bakterien, Insekten oder Mammalia entwickelten sich Verteidigungssysteme, die eine Infektion der entsprechenden Viren gänzlich verhindern oder unterbrechen. Viren wiederum gelang es mit der Zeit diese antiviralen Verteidigungssysteme zu umgehen. Dies wird vor allem durch das Einbringen viraler Phosphodiesterasen (PDEs) erreicht. Phosphodiesterasen besitzen aufgrund ihrer zentralen Position bei vielen Virusinfektionen pharmakologische Relevanz. Diese Enzyme und/oder ihre Inhibitoren könnten Kandidaten für antivirale sowie antibakterielle Wirkstoffe darstellen. Im Institut für Pharmakologie werden daher Phosphodiesterasen verschiedener Viren identifiziert und deren enzymatische Aktivität untersucht. Im Folgenden wird die Funktion von Phosphodiesterasen bei Virusinfektionen an zwei Beispielen erläutert. Diese sind auch Teil der Forschung unseres Instituts.
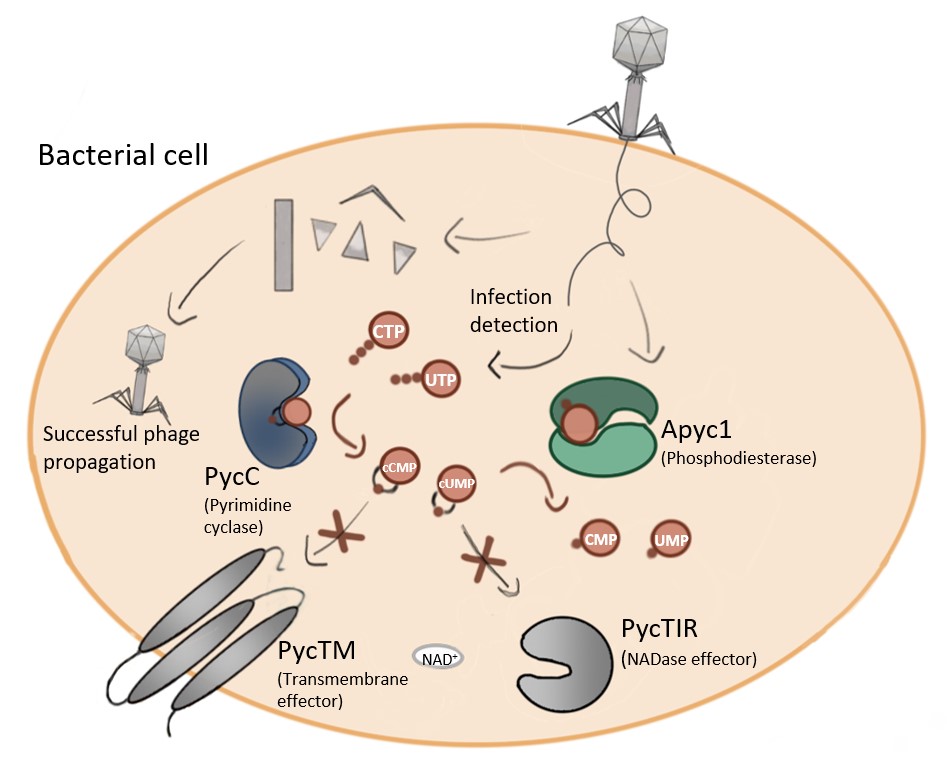
- Apyc1 – eine Phosphodiesterase aus Bakteriophagen
In Bakterien entwickelten sich verschiedene Abwehrsysteme, um sich gegen eine Phageninfektion zu wehren. Das Abwehrsystem Pycsar (pyrimidine cyclase system for anti-phage resistance) beruht auf der Aktivierung der Wirtsimmunität durch die Bildung von zyklischen Nukleotiden. Dabei handelt es sich um die Pyrimidinnukleotide cCMP und cUMP. Diese aktivieren anschließend bestimmte Effektoren, was zum Absterben der Bakterienzellen führt. Die Vermehrung der Phagen wird so verhindert und die Bakterienpopulation geschützt. Einige Phagen wiederum kodieren für eine Phosphodiesterase namens Apyc1, die cCMP und cUMP spaltet, und so das bakterielle Verteidigungssystem außer Kraft setzt.
- Poxine – Nukleasen aus humanpathogenen Viren
Auch humanpathogene Viren entwickelten Strategien, um die zelluläre Immunabwehr zu umgehen. Zum Beispiel exprimieren Pockenviren, Baculoviren und Gelbfieberviren Nukleasen, die zyklische Dinukleotide schneiden. Diese Nukleasen werden Poxine (poxvirus immune nucleases) genannt und kommen unter anderem im Vaccina- oder Mpox-Virus vor. Sie sind in der Lage cGAMP zu spalten und ermöglichen somit das Umgehen des zellulären Signalweg cGAS-STING, der zum Immunsystem gehört.
Mögliche Themen für experimentelle Arbeiten
(Bachelor- und Masterarbeiten, Laborpraktika):
- Screening von relevanten Virusgenomen auf Gene, die für PDE-/Nukleasehomologe kodieren und Produktion/Reinigung der entsprechenden Enzyme
- Identifizierung von optimalen Reaktionsbedingungen für PDEs/Nukleasen
- Kinetische Charakterisierung der cNMP-hydrolytischen Aktivität von PDEs/Nukleasen
- Screening nach Wirkstoffen, die die cNMP-hydrolytischen Aktivität von PDEs/Nukleasen modulieren
Methodenspektrum
Zusätzlich zu den grundlegenden molekularbiologischen, immunologischen, Proteinbiochemischen und zellbiologischen Labortechniken:
- Design von Expressionsvektoren
- Transformation und Kultivierung von Bakterien
- High-throughput Spektralphotometrie/-fluorimetrie/-luminometrie
- ELISA, FRET, BRET
- Küvetten-Spektralphotometrie
- Chromatographische Präparation und Analyse von Proteinen (ÄKTA)
- Protein-Analyse
- SDS-PAGE
- Western-Blot (ECL)
- LC-IMS/QTOF, LC-MS/MS, GC-MS/MS (Kooperation mit RCU Metabolomics)
Ausgewählte Publikationen:
Seifert and Schirmer (2021). cCMP and cUMP come into spotlight , finally. Trends in biochemical sciences, 47(6), 461-463. https://doi.org/10.1016/j.tibs.2021.12.008
Seifert and Bugert (2023). cCMP and cUMP phosphodiesterases in viral infections. Trends in biochemical sciences, 48(10), 835-838. https://doi.org/10.1016/j.tibs.2023.05.013
Nähere Informationen:
Dr. Lina Schütte, E-Mail