Projektvorstellung 2023
Single-cell transcriptomics reveals common epithelial response patterns in human acute kidney injury
Christian Hinze, Christine Kocks, Janna Leiz, Nikos Karaiskos, Anastasiya Boltengagen, Shuang Cao, Christopher Mark Skopnik, Jan Klocke, Jan-Hendrik Hardenberg, Helena Stockmann, Inka Gotthardt, Benedikt Obermayer, Laleh Haghverdi, Emanuel Wyler, Markus Landthaler, Sebastian Bachmann, Andreas C Hocke, Victor Corman, Jonas Busch, Wolfgang Schneider, Nina Himmerkus, Markus Bleich, Kai-Uwe Eckardt, Philipp Enghard, Nikolaus Rajewsky, Kai M Schmidt-Ott
Eine akute Nierenschädigung ist eine ernstzunehmende Komplikation und betrifft ca. 20% in Krankenhäusern behandelter Patientinnen und Patienten. Bei intensivmedizinisch betreuten Patientinnen und Patienten erleiden sogar bis zu 50% der Behandelten eine akute Nierenschädigung. Die Ursachen hierfür sind sehr heterogen, ein akute Nierenschädigung kann z. B. durch schwere Traumata und Infektionen, komplizierte Operationen, sowie Intoxikationen und unerwünschte Arzneimitteleffekte ausgelöst werden. Das Vorliegen selbst einer milden akuten Nierenschädigung ist mit einer erhöhten Sterblichkeit im weiteren Langzeitverlauf assoziiert. Leider existieren für die meisten Formen der akuten Nierenschädigung heutzutage keine gezielten, auf die Niere gerichteten Therapien. Ursache hierfür ist nicht zuletzt ein sehr unvollständiges Verständnis der genauen molekularen Vorgänge in den Nieren im Falle einer akuten Nierenschädigung.
Im Forschungsbericht 2023 stellen wir ihnen deshalb eine Arbeit der Nephrologen Dr. Christian Hinze und Professor Kai Schmidt-Ott aus der Klinik für Nieren- und Hochdruckerkrankungen vor, die in Zusammenarbeit mit Wissenschaftlerinnen und Wissenschaftlern der Charité-Universitätsmedizin und des Max-Delbrück Centrums in Berlin den molekularen Vorgängen bei akuter Nierenschädigung nachgegangen sind. Die zugrundeliegende Arbeit wurde in der Zeitschrift Genome Medicine veröffentlicht.
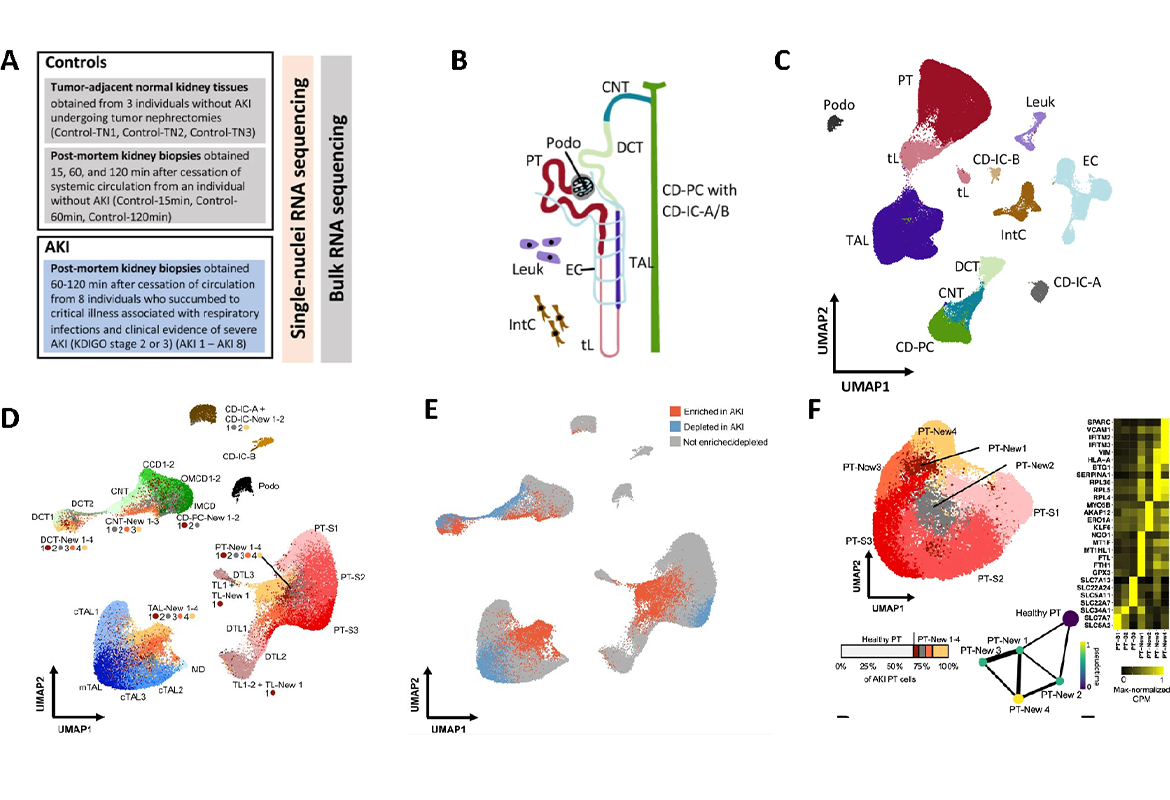
Da die Niere selbst aus einer Vielzahl verschiedener Zelltypen besteht, nutzten die Forschenden hochauflösende molekulare Verfahren der Einzelzellsequenzierung, die es ermöglichen, das Genexpressionsprofil tausender Zellen individuell zu untersuchen. Nierenbiopsien wurden von intensivmedizinisch wegen schwerer Atemwegsinfekte betreuten Patientinnen und Patienten mit schwerer akuter Nierenschädigung gewonnen. Diese wurden gegen gesundes Kontrollnierengewebe verglichen, welches zum Beispiel bei Nierenoperationen aus nicht erkrankten Bereichen der Nieren gewonnen wurde.
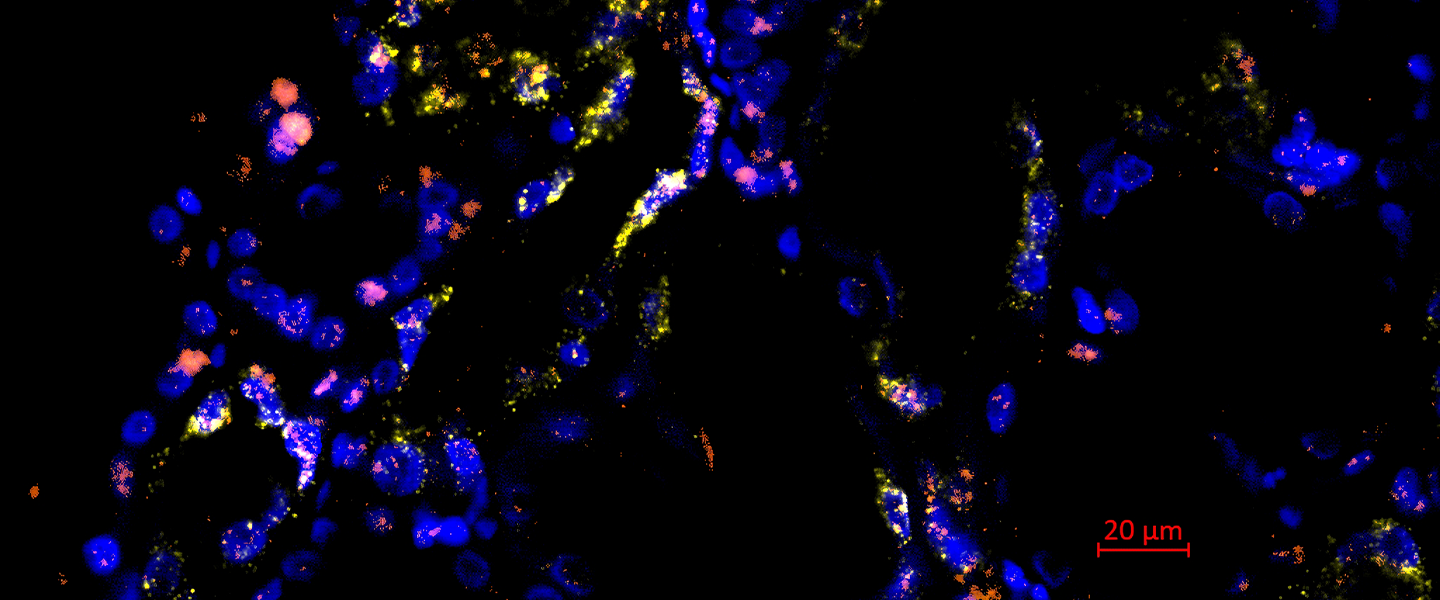
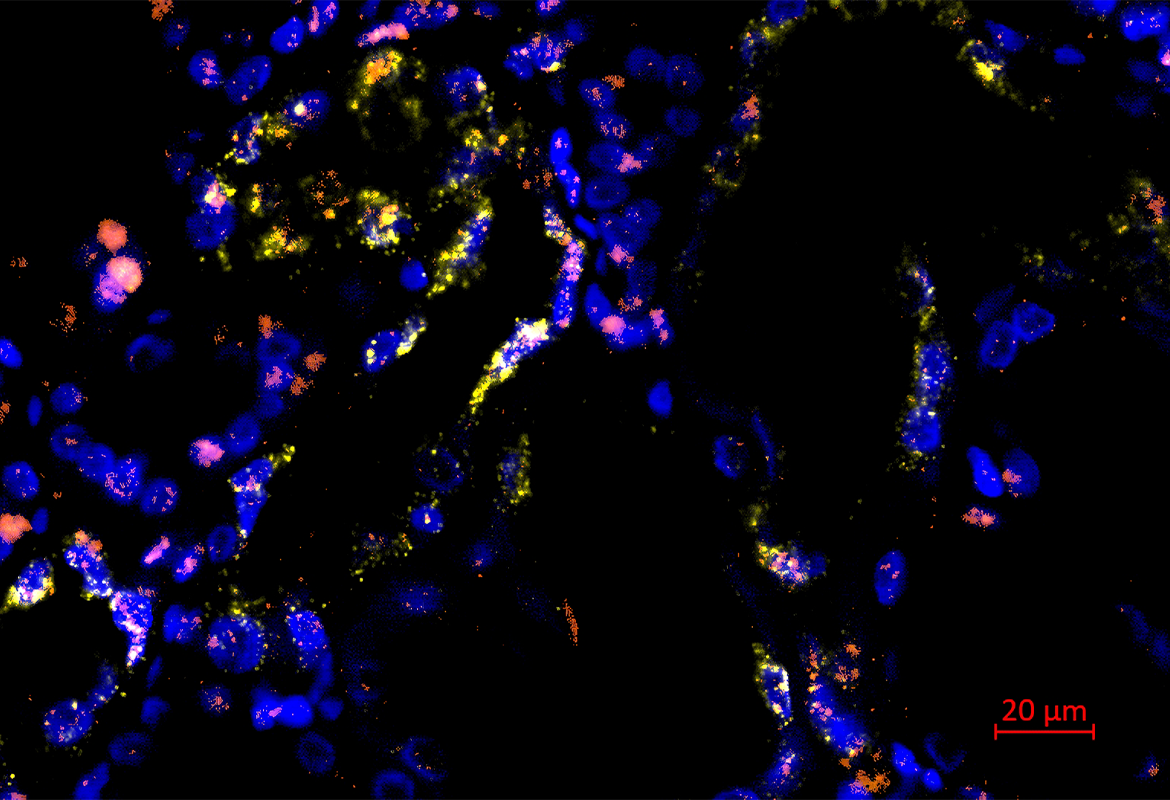
Für diese Arbeit sequenzierten die Forschenden das Transkriptom (also die Gesamtheit der in einer Zelle abgeschriebenen Gene) von mehr als 100000 individuellen Zellen von insgesamt 12 Patientinnen und Patienten (8 mit akuter Nierenschädigung und 4 Kontrollen ohne akute Nierenschädigung). Bioinformatische Analysen ermöglichten die Zuordnung der so sequenzierten Zellen zu den ursprünglichen Zelltypen. Im Rahmen der Analyse fiel auf, dass sich die Genexpression der von akuter Nierenschädigung Betroffenen erheblich von der des gesunden Nierengewebes unterschied. So sah man, dass eine akute Nierenschädigung zur Bildung neuer krankhafter Zellzustände in Zellen des Nierentubulussystems führte. Interessanterweise schienen diese Veränderungen in unterschiedlichen Zelltypen der Nierentubuli auf ähnliche Weise abzulaufen. So konnten die Forschenden vier verschiedene Schädigungs-assoziierte Zellzustände identifizieren, die jeweils definierte Genexpressionsprofile aufwiesen und durch verschiedene Methoden validiert werden konnten. Neben Unterschieden im Genexpressionsprofil unterschieden sich die mit akuter Nierenschädigung assoziierten Zellzustände offenbar auch hinsichtlich ihres Potentials wieder zu gesunden Nierenzellen zu regenerieren, was ein bioinformatischer Abgleich mit zeitlich aufgelösten Einzelzellsequenzierungsdaten aus einem Mausmodell der Nierenschädigung zeigte. Einige der Schädigungs-assoziierten Zellen scheinen das Potential zu haben, sich wieder zu gesundem Nierengewebe zu entwickeln, während andere Zellen ein irreversibles Schädigungsmuster aufwiesen (siehe Abbildung).
Die Arbeit zählt zu einer der ersten Studien, die mittels Einzelzellsequenzierung die Vielfalt der zelltypspezifischen Genexpressionsantworten auf akute Nierenschädigung beim Menschen untersucht hat. Die Signalwege, die in den Schädigungs-assoziierten Zellen aktiviert werden, geben neue Anhaltspunkte für mögliche therapeutische Interventionen. So könnten neue Therapien darauf abzielen, die für irreversibel geschädigte Zellen typischen Signalwege zu hemmen.
Publikation
Single-cell transcriptomics reveals common epithelial response patterns in human acute kidney injury
Hinze C, Kocks C, Leiz J, Karaiskos N, Boltengagen A, Cao S, Skopnik CM, Klocke J, Hardenberg JH, Stockmann H, Gotthardt I, Obermayer B, Haghverdi L, Wyler E, Landthaler M, Bachmann S, Hocke AC, Corman V, Busch J, Schneider W, Himmerkus N, Bleich M, Eckardt KU, Enghard P, Rajewsky N, Schmidt-Ott KM
Weitere Informationen
Herausgeber:
Präsident der Medizinischen Hochschule Hannover
Herr Prof. Dr. med. Michael P. Manns
Forschungsdekan der Medizinischen Hochschule Hannover
Herr Prof. Dr. med. Frank M. Bengel
Bearbeitung und Ansprechpartner:
Berichtswesen der Medizinischen Hochschule Hannover
Alica Wollmann
Telefon: 0511/ 532– 5578
E-Mail: wollmann.alica@mh-hannover.de
Online Umsetzung:
Forschungsdekanat der Medizinische Hochschule Hannover
Jan Tauwaldt
und
Berichtswesen der Medizinischen Hochschule Hannover
Alica Wollmann
Telefon: 0511/ 532- 5578
Forschungsbericht 2023
Hier finden Sie den mit Hilfe des Forschungsinformationssystems (FIS) erstellten Forschungsbericht. Wie in den vergangenen Jahren möchten wir hier die Gelegenheit nutzen, explizit ein Projekt stellvertretend vorzustellen.
Forschungsinformationssystem (FIS)Hier finden Sie weitere Informationen zum Forschungsinformationssystem (FIS).
Hochschulbibliographie- Projektvorstellung 2022
- Projektvorstellung 2021
- Projektvorstellung 2020
- Projektvorstellung 2019
- Projektvorstellung 2018
- Projektvorstellung 2017
- Forschungsbericht 2016
- Forschungsbericht 2015
- Forschungsbericht 2014
- Forschungsbericht 2013
- Forschungsbericht 2012
- Forschungsbericht 2011
- Forschungsbericht 2010
- Forschungsbericht 2009
- Forschungsbericht 2008
- Forschungsbericht 2007